修正 C++ 的程式碼在使用一定量動態記憶體後會產生 RF 的問題 @ 2019/12/6 4:45pm
Submit Ranklist
Problem : 232 - DNA
Problem Statistics
Solved Member: 10 Submission: 32 User Tried: 10Statement:
電腦的一項有趣的用途便是用來分析生物學的資料諸如 DNA 序列。就生物學而言,一股的 DNA 是由一連串的腺嘌呤(Adenine)、胞嘧啶(Cytosine)、鳥嘌呤(Guanine)、胸腺嘧啶(Thymine)等四種核甘酸(nucleotide)組成。這四種核甘酸將以字元 A、C、G 及 T 來分別表示。因此,一股的DNA 可以用這四個字元的一連串組合來表示。我們稱這樣的字元串組合為一串 DNA 序列。
生物學家不能夠確定一串 DNA 序列中某些核甘酸的情況是有可能會發生的。在種情下,可以用字元 N 來表示出現在 DNA 序列中某一位置的未知核甘酸。換句話說,N 可以視為一個萬用字元而用來表示A、C、G 或T 其中的任一個字元。若一串 DNA 序列中含有一個或更多的字元N 就稱作「不完整序列」;否則,稱作「完整序列」。一串完整序列會被認為與另一串完整序列吻合,是發生在當它可以是經由以四種核甘酸中的任一個來取代不完整序列中的每個N 而得的結果。例如 ACCCT 與 ACNNT 是吻合的,但 AGGAT 則是不吻合的。
研究人員往往會照依英文字母順序來排序這四種核甘酸: A 在 C 之前, C 在 G 之前, G 在 T 之前。一串DNA 序列將被分類成 「類型1」,如果此序列中每一個核甘酸與右邊緊鄰核甘酸是相同或者其字母順序是在右邊緊鄰核甘酸的字母順序之前。例如 AACCGT 是類型1,但 AACGTC 則不是。
通常一串類型 j 序列(j > 1)可以是一串類型(j-1)序列,或者是由一串類型(j-1)序列與一串類型1 序列串接而成的。例如 AACCC、ACACC 與 ACACA 是類型3 序列,但 GCACAC 與 ACACACA 則不是。
再者,研究人員也會依照英文辭典中詞的編纂順序方式來排序 DNA 序列。據此,第1個長度為5的類型3序列將是 AAAAA,最後一個是 TTTTT。另一個例子是,當考慮一串不完整序列ACANNCNNG 時,前7串與它吻合的類型3序列是:
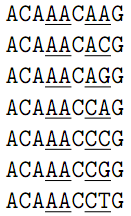
生物學家不能夠確定一串 DNA 序列中某些核甘酸的情況是有可能會發生的。在種情下,可以用字元 N 來表示出現在 DNA 序列中某一位置的未知核甘酸。換句話說,N 可以視為一個萬用字元而用來表示A、C、G 或T 其中的任一個字元。若一串 DNA 序列中含有一個或更多的字元N 就稱作「不完整序列」;否則,稱作「完整序列」。一串完整序列會被認為與另一串完整序列吻合,是發生在當它可以是經由以四種核甘酸中的任一個來取代不完整序列中的每個N 而得的結果。例如 ACCCT 與 ACNNT 是吻合的,但 AGGAT 則是不吻合的。
研究人員往往會照依英文字母順序來排序這四種核甘酸: A 在 C 之前, C 在 G 之前, G 在 T 之前。一串DNA 序列將被分類成 「類型1」,如果此序列中每一個核甘酸與右邊緊鄰核甘酸是相同或者其字母順序是在右邊緊鄰核甘酸的字母順序之前。例如 AACCGT 是類型1,但 AACGTC 則不是。
通常一串類型 j 序列(j > 1)可以是一串類型(j-1)序列,或者是由一串類型(j-1)序列與一串類型1 序列串接而成的。例如 AACCC、ACACC 與 ACACA 是類型3 序列,但 GCACAC 與 ACACACA 則不是。
再者,研究人員也會依照英文辭典中詞的編纂順序方式來排序 DNA 序列。據此,第1個長度為5的類型3序列將是 AAAAA,最後一個是 TTTTT。另一個例子是,當考慮一串不完整序列ACANNCNNG 時,前7串與它吻合的類型3序列是:
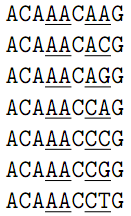
Task:
寫一個程式,當給定一串長度為 M 的不完整序列後,尋找與它吻合的第 R 串類型 K 序列。
Input:Output:
輸入第一行有 3 個整數以空白鍵分隔:M (1 ≤ M ≤ 50,000)、K (1 ≤ K ≤ 10) 和 R (1 ≤ R ≤ 2 × 1012)。第二行包含一串長度為 M 的字元序列,代表不完整序列。
與此串不完整序列吻合的類型 K 序列的個數將不會大於 4 × 1018。此外, R 不會大於與所給定的不完整序列吻合之類型 K 序列的個數。
在共20分的測試資料組別中,M 最多為10。
與此串不完整序列吻合的類型 K 序列的個數將不會大於 4 × 1018。此外, R 不會大於與所給定的不完整序列吻合之類型 K 序列的個數。
在共20分的測試資料組別中,M 最多為10。
在第一行列印出與輸入的不完整序列吻合的第 R 串類型 K 序列。
Sample Input:Sample Output:
Sample Input 1
9 3 5
ACANNCNNG
Sample Input 2
5 4 10
ACANN
9 3 5
ACANNCNNG
Sample Input 2
5 4 10
ACANN
Sample Output 1
ACAAACCCG
Sample Output 2
ACAGC
ACAAACCCG
Sample Output 2
ACAGC
Source:
APIO 2008Problem Setter
hanhan0912Testdata:
| Test | Time | Memory | Score |
|---|---|---|---|
| 0-1 | 1000ms | 131072kb | |
| 0-2 | 1000ms | 131072kb | |
| 1 | 1000ms | 131072kb | 5 |
| 2 | 1000ms | 131072kb | 5 |
| 3 | 1000ms | 131072kb | 5 |
| 4 | 1000ms | 131072kb | 5 |
| 5 | 2000ms | 131072kb | 7 |
| 6 | 2000ms | 131072kb | 7 |
| 7 | 2000ms | 131072kb | 7 |
| 8 | 2000ms | 131072kb | 7 |
| 9 | 2000ms | 131072kb | 7 |
| 10 | 2000ms | 131072kb | 7 |
| 11 | 2000ms | 131072kb | 8 |
| 12 | 2000ms | 131072kb | 7 |
| 13 | 2000ms | 131072kb | 8 |
| 14 | 2000ms | 131072kb | 7 |
| 15 | 2000ms | 131072kb | 8 |
NeoHOJ — The new era of HOJ
頁面讀取時間: 0.0073 秒,使用記憶體: 0.46MB 。
GitHub